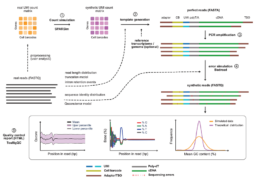
AsaruSim: a single-cell and spatial RNA-Seq Nanopore long-reads simulation workflow
AsaruSim est un workflow open-source développé par la plateforme génomique GenomiqueENS de l’IBENS (Institut de Biologie de l’École normale supérieure), qui permet de simuler des datasets Nanopore synthétiques à lecture longue sur cellule unique, en reproduisant fidèlement les données expérimentales réelles. AsaruSim utilise un processus en plusieurs étapes qui comprend la création d’une matrice de comptage synthétique, la génération de reads idéaux, l’amplification PCR optionnelle, l’introduction d’erreurs de séquençage et un rapport complet sur le contrôle de la qualité. Appliqué à un ensemble de données de cellules sanguines mononucléaires, AsaruSim a reproduit avec précision les caractéristiques de lecture expérimentales.
Les utilisations potentielles incluent la génération de lectures avec expression différentielle des gènes ou DEI entre les groupes de cellules, ainsi que la simulation de changements de seuils connus ou d’effets de lots, afin d’évaluer et d’optimiser les méthodes single-cell long-read .
Actuellement, AsaruSim génère des données compatibles avec les protocoles 10X Genomics 3ʹ et spatiale transcriptomique.

Le code source d’AsaruSim est disponible sur GitHub :
https://github.com/GenomiqueENS/asarusim
Contact : hamraoui@bio.ens.psl.eu