Approches à l’échelle du transcriptome ou basées sur le séquençage
I) Les méthodes plus anciennes se concentraient sur l’acquisition ciblée de sous-ensembles d’échantillons pour le séquençage et utilisaient la microscopie à capture laser (LCM) et des méthodes à marqueurs photo-clivables, où des régions spécifiques marquées par des marqueurs de surface ou des sondes d’ARNm étaient collectées et séquencées. Ces technologies permettent un profilage dirigé par l’utilisateur de régions d’intérêt spécifiques (ROIs) avec aussi peu que 10 cellules, ce qui permet aux chercheurs de caractériser de multiples répliques ou types/de localisations tissulaires pour chaque échantillon [1].
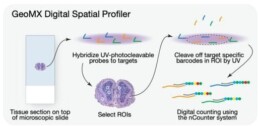
-> NanoString GeoMx DSP est un exemple contemporain de cette technologie.
Le NanoString GeoMx Digital Spatial Profiler (DSP) est une plate-forme de profilage spatial de nouvelle génération qui permet aux chercheurs d’analyser l’expression génique, l’expression protéique et la morphologie cellulaire in situ, ou dans leur contexte tissulaire natif. La plateforme GeoMx DSP est basée sur une technologie de marquage numérique qui permet un profilage à haut débit des cibles ARN et protéiques dans des régions d’intérêt définies spatialement à l’intérieur d’échantillons tissulaires [2].
La plate-forme utilise un ensemble d’oligonucléotides à code-barres d’ADN, appelés sondes, conçues pour cibler des molécules d’ARN ou de protéines spécifiques d’intérêt. Ces sondes sont hybridées à la section de tissu, et le motif d’hybridation résultant est lu à l’aide d’un système d’imagerie numérique. Cette approche permet la détection simultanée de milliers de cibles ARN ou protéiques dans une seule section de tissu.

II) Les méthodes qui permettent une résolution au niveau de la cellule unique ou subcellulaire reposent sur le marquage spatial et le séquençage in situ. Ces technologies utilisent des nano-billes en forme de grille ou des sites de séquençage sur la lame [1].
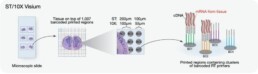
-> 10X Visium pour la version multi-cellulaire de la même technologie.
10X Genomics Visium est une technologie de transcriptomique spatiale qui permet l’analyse simultanée de l’expression génique et de l’information spatiale dans des échantillons de tissus intacts. Cette technologie permet aux chercheurs de cartographier le transcriptome des cellules individuelles jusqu’à leur emplacement spécifique dans le tissu, offrant ainsi des informations sur l’organisation spatiale de l’expression génique.
Le processus de Visium implique la préparation d’échantillons de tissus frais ou fixés en sections minces, suivie de la perméabilisation des sections de tissus et de l’hybridation de sondes oligonucléotidiques codées en barres pour capturer les molécules d’ARN. Les molécules d’ARN capturées sont ensuite séquencées et les données résultantes sont traitées à l’aide d’un logiciel propriétaire pour générer des profils d’expression génique résolus spatialement.
L’une des caractéristiques clés de la technologie Visium est sa capacité à analyser des échantillons de tissus sans avoir besoin de microdissection ou de dissociation, ce qui peut perturber l’organisation spatiale des cellules et de l’expression génique. Cela permet d’analyser des structures tissulaires complexes, telles que le cerveau, où les interactions cellule-cellule et les relations spatiales sont critiques pour comprendre la fonction et les maladies.
[1] Park, J., Kim, J., Lewy, T. et al. Spatial omics technologies at multimodal and single cell/subcellular level. Genome Biol 23, 256 (2022). https://doi.org/10.1186/s13059-022-02824-6
[2] Zollinger DR, Lingle SE, Sorg K, Beechem JM, Merritt CR. Geomx™ RNA assay: high multiplex, digital, spatial analysis of RNA in FFPE tissue. Methods Mol Biol. 2020 ; 2148:331–45.

