Transcriptomique spatiale
La contribution de l’analyse à cellule unique est majeure pour l’interprétation biologique. Cependant, l’organisation des tissus dans l’espace reste une information essentielle (surtout dans l’étude des échantillons tumoraux) qui n’est pas résolue par les techniques conventionnelles en cellule unique.
–>Mise en place de technologies de transcriptomique spatiale.
La transcriptomique spatiale est une technologie qui combine le profilage à haut débit de l’expression des gènes avec l’information spatiale des cellules dans les tissus ou les organes. Elle permet aux chercheurs d’identifier et de cartographier les motifs d’expression génique des cellules individuelles dans leur contexte tissulaire natif, offrant ainsi une compréhension plus complète de l’organisation moléculaire et cellulaire des tissus.
En transcriptomique traditionnelle, l’ARN est extrait d’un tissu ou d’une population de cellules et séquencé pour déterminer le profil d’expression génique de l’échantillon. Cependant, cette approche ne fournit pas d’informations sur la distribution spatiale des gènes exprimés dans le tissu. La transcriptomique spatiale permet de surmonter cette limitation en préservant physiquement l’arrangement spatial des cellules dans le tissu au cours du processus de séquençage.
En RNA-seq cellule unique, les chercheurs dissocient les cellules du tissu pour, par exemple, distinguer les types de cellules sur la base de l’expression génique. Maintenant, avec les méthodes de transcriptomique résolues spatialement, les scientifiques peuvent obtenir des données transcriptomiques et connaître le contexte positionnel de ces cellules dans un tissu [1].
Actuellement, il existe plus de 50 technologies de cartographie spatiale différentes disponibles.
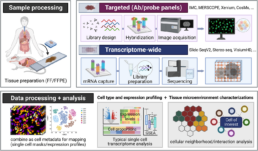
Ces dernières années, les méthodes expérimentales traditionnelles, telles que le marquage avec des reporteurs, l’immunohistochimie (IHC) et l’hybridation in situ fluorescente (FISH), ont laissé place aux technologies d’omique spatiale pour couvrir un plus grand nombre de transcrits ou de zones (Fig. 1). De manière générale, les technologies d’omique spatiale varient en termes de résolution spatiale (taille minimale des unités moléculaires profilées), de couverture (étendue des tissus couverts), d’échelle et de débit (nombre d’échantillons et vitesse de profilage) et de capacité de multiplexage (étendue des entités moléculaires profilées simultanément).
En fonction de la question de recherche, les méthodes de profilage peuvent être divisées en I) approches ciblées ou multiplexées à base de sondes ou d’anticorps et II) approches à l’échelle du transcriptome ou basées sur le séquençage de nouvelle génération (NGS). La plupart des technologies d’omique spatiale avec une résolution au niveau subcellulaire sont effectuées sur lame (in situ) en utilisant soit la microscopie soit du séquençage haut débit [2].
[1] Marx, V. Method of the Year: spatially resolved transcriptomics. Nat Methods 18, 9–14 (2021). https://doi.org/10.1038/s41592-020-01033-y
[2] Park, J., Kim, J., Lewy, T. et al. Spatial omics technologies at multimodal and single cell/subcellular level. Genome Biol 23, 256 (2022). https://doi.org/10.1186/s13059-022-02824-6