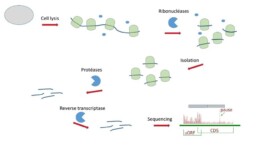
Ribo Seq
RiboSeq est utilisé pour cartographier et quantifier les empreintes sur les transcrits (~30nt) des ribosomes fonctionnels à un temps donné dans une cellule.
Le protocole consiste à d’abord figés les ribosomes sur l’ARNm grâce à des inhibiteurs de traduction. Ensuite l’échantillon est traité avec des ribonucléases pour dégrader tous les ARNm qui ne sont pas protégés par un ribosome. Les complexes ARNm-ribosome sont isolés. Après traitement avec des protéases, les fragments d’ARNm « protégés » sont purifiés, transcrits en ADNc et séquencés.
L’identification de ces régions permet de décrire l’occupation des ribosomes et l’efficacité de translation des ARNm (ratio entre comptage des empreintes RiboSeq et comptage de copie RNAseq d’un ARNm donné), les sites alternatifs de transcription, des sites de pause la translation, expression différentielle des gènes post transcription etc. ( revues: Ingolia N.T et al. 2018 ; Kage U et al. 2020)