Cartographie des sites d’initiation de la transcription : TSS seq
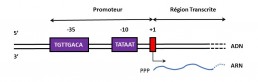
Le site d’initiation de la transcription (en anglais, transcription start site, TSS) est le premier nucléotide de l’ADN qui est transcrit en ARN (Fig1). Il est difficile de déterminer sa position exacte par bioinformatique, mais il existe plusieurs méthodes expérimentales pour le localiser, notamment le séquençage à haut débit.
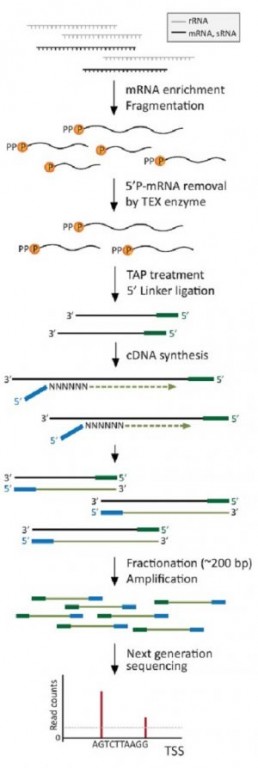
Le protocole de cartographie des TSS utilise la TAP ou « Tobacco Acid Pyrophosphatase », enzyme permettant la conversion des extrémités 5’PPP des ARNs natifs en extrémités 5’P, compatibles avec la ligation d’adaptateurs (Fig2).
L’étude est réalisée en comparant les résultats de séquençage des extrémités 5’ des ARN traités ou non par la TAP. Sans traitement, le séquençage donnera accès à tous les ARNs (ARN messagers en cours de dégradation ou matures) à l’exception de ceux dont l’extrémité 5’ est native. Après traitement à la TAP, les mêmes séquences seront obtenues, avec en plus les séquences des ARNs natifs (S Cho et al.) .
Plateformes à contacter pour ce domaine d’expertise
Rubriques associées
- Séquençage des petits ARN
- TAPS/TAPSβ
- Enzymatic Methyl-seq (EM-seq™)
- Méthylation de l’ADN et ARN Natifs
- Cartographie des sites ADN – Protéines de la chromatine : CUT & RUN vs CUT & Tag
- Cartographie des contacts de la chromatine : 3C, 4C et 5C
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie des sites d’accessibilité de la chromatine : DNase-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : FAIRE seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq
- BiSeq