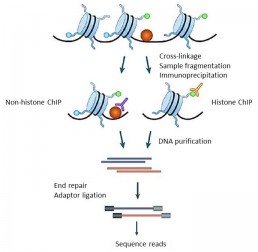
Cartographie des sites des interactions ADN- protéines : CHIP seq
Chromatin ImmunoPrecipitation Sequencing est une méthode utilisée pour analyser les interactions entre les protéines et l’ADN. Cette technologie combine l’immunoprécipitation de la chromatine (ChIP), en utilisant des anticorps spécifiques d’une protéine d’intérêt et le séquençage haut débit.
Le ChIP seq permet de cartographier tous les sites de liaison sur l’ADN d’une protéine d’intérêt à l’échelle du génome. Par exemple, il est possible d’identifier toutes les régions génomiques associées avec une histone nucléosomale portant une modification biochimique (acétylation, méthylation, etc.) caractéristique de l’activité transcriptionnelle ou encore de définir les sites de liaison de facteurs de transcription (PJ Park et al. 2009).
Le principe de la méthode consiste à fixer de façon covalente les protéines liées à l’ADN par un traitement chimique, de fragmenter la chromatine, d’isoler des complexe ADN-protéines par immunoprécipitation en présence de l’anticorps d’intérêt. Une fois l’élimination des protéine et la purification, les fragments d’ADN obtenus sont séquencés.
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- TAPS/TAPSβ
- Enzymatic Methyl-seq (EM-seq™)
- Méthylation de l’ADN et ARN Natifs
- Cartographie des sites ADN – Protéines de la chromatine : CUT & RUN vs CUT & Tag
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq
- BiSeq