Cartographie des contacts cis et trans de la chromatine : HiC-seq
L’étude de la conformation 3D de la chromatine et des régions d’interaction cis (intra chromosome) et trans (inter chromosome) permet l’identification de domaines topologiques (TADs). Les TADs ne sont pas que des compartiments structuraux de la chromatine mais aussi des régions de réplication de l’ADN et de régulation du génome (Lupiáñez et al. 2015).
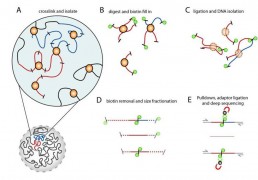
La méthode HiC-seq est basée sur l’immobilisation de la conformation de la chromatine et le séquençage des régions d’interaction, ce qui permet d’avoir une image de l’activité des TADs à un temps donné.
La chromatine est figée dans sa conformation 3D avec du formaldéhyde (crosslinking). L’ADN est fragmenté par digestion enzymatique (HindIII), les extrémités cohésives des sites de restriction sont comblées avec des nucléotides biotinilés. Après ligation des nouvelles extrémités franches, une purification sur billes streptavidine permet d’isoler spécifiquement l’ADN des régions d’interactions.
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- TAPS/TAPSβ
- Enzymatic Methyl-seq (EM-seq™)
- Méthylation de l’ADN et ARN Natifs
- Cartographie des sites ADN – Protéines de la chromatine : CUT & RUN vs CUT & Tag
- Cartographie des contacts de la chromatine : 3C, 4C et 5C
- Cartographie des sites d’accessibilité de la chromatine : DNase-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : FAIRE seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq
- BiSeq