BiSeq
La méthylation de l’ADN est l’une des modifications épigénétiques les plus étudiées car elle joue un rôle important dans la régulation de l’expression génétique et par conséquence dans de nombreux processus biologiques et dans un grand éventail de pathologies.
Grâce aux technologies de séquençage de nouvelle génération (NGS) l’étude du méthylome est possible sur le génome entier avec une résolution au niveau de la paire de bases .
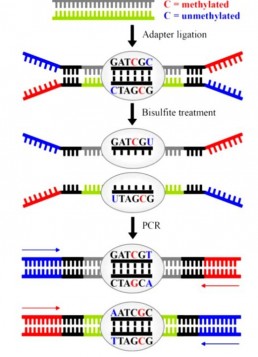
La technique BiSeq (Bisulfite sequencing) repose sur le traitement de l’ADNsb au bisulfite de sodium qui désamine les C non methylée (C) plus rapidement que les C méthylées (5mC et 5hmC). Ce traitement qui converti les C en Uracil puis en T après amplification (Fig1) permet de transformer la marque épigénétique en variation génétique identifiable par séquençage (WS Yong et al. 2016) .
Le protocole BiSeq consiste à préparer des fragments d’ADN par sonication , les indexer avec des adaptateurs methylés puis les sélectionner par la taille. Les banques sont ensuite traités au bisulfite de sodium, amplifiés et séquencés. La réaction de conversion peut être vérifiée en intégrant un ADN contrôle non méthylé, sachant qu’un temps d’incubation excessif ou qu’un mauvais ration bisulfite/ADN non seulement dénature l’ADN mais surtout peut modifier les C méthylées avec pour résultat une sous-estimation de l’état de méthylation de l’échantillon (PW Laird. 2010).
L’analyse de données BiSeq est complexe par le fait que la conversion crée une transition C=>T sur chaque position de chacun des brins d’ADN. Pour chaque locus séquencé devront être alignés le brin « riche » en G, son complémentaire « riche » en C, le brin converti « riche » en A et son complémentaire « riche » en T (Fig1). La méthode « BiSeqS » (AK Mattox et al. 2017) introduit un barcodage moléculaire pour discriminer l’état de méthylation de chacun des 2 brins d’ADN et simplifier l’analyse.
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- TAPS/TAPSβ
- Enzymatic Methyl-seq (EM-seq™)
- Méthylation de l’ADN et ARN Natifs
- Cartographie des sites ADN – Protéines de la chromatine : CUT & RUN vs CUT & Tag
- Cartographie des contacts de la chromatine : 3C, 4C et 5C
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie des sites d’accessibilité de la chromatine : DNase-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : FAIRE seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq