Le séquençage du Regulome
Le régulome fait référence à l’interaction entre l’ensemble des composants régulateurs d’une cellule qui contrôlent l’état d’expression des gènes et de leurs isoformes, en fonction de leur localisation subcellulaire, de leur état tissulaire, de leur développement et de leur pathologie.
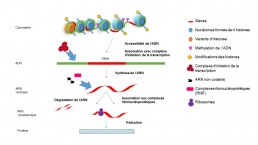
La régulation peut s’exercer à plusieurs étapes (Fig1.).
1- L’accessibilité de l’ADN pour la transcription des gènes dépend de facteurs qui peuvent modifier le compactage des 5 familles d’histones sur lesquelles s’enroule l’ADN (Tsompana & Buck 2014; Klein & Hainer 2020; Sati &Cavalli 2016).
2- Le niveau de méthylation de l’ADN (ajout/suppression de groupements CH3 sur la Cytosine) module la transcription des gènes.
3- Les protéines régulatrices (facteurs de transcription) ont la capacité de se lier de façon spécifique à de courtes séquences de DNA et de réguler de façon positive ou négative la transcription d’un gène.
4- Des interactions ARN-protéines (RNP : complexes ribonucléoprotéiques) agissent sur les mécanismes post-trancriptionnels comme l’épissage, stabilité des ARNs etc.
5- Différentes classes d’ARN non codants comme les petits et micro ARN peuvent intervenir dans l’épissage, la synthèse des ribosomes, la dégradation les ARNm, la répression de la traduction.
Tous ces mécanismes sont affectés par des facteurs environnementaux (alimentation, pollution, âge, stress, traitement médicamenteux etc.).
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- TAPS/TAPSβ
- Enzymatic Methyl-seq (EM-seq™)
- Méthylation de l’ADN et ARN Natifs
- Cartographie des sites ADN – Protéines de la chromatine : CUT & RUN vs CUT & Tag
- Cartographie des contacts de la chromatine : 3C, 4C et 5C
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie des sites d’accessibilité de la chromatine : DNase-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : FAIRE seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq
- BiSeq