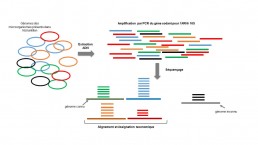
Métagénomique sur amplicon ciblé/barcoding/metabarcoding
Les études génomique pour la détermination de la composition d’un microbiome en terme d’espèce, consistent à séquencer un ou plusieurs gènes particuliers appelés barcodes/ étiquettes, commun à plusieurs espèces. Comme exemple, le séquençage ciblé du gène codant l’ARNr 16S mettra en évidence la représentation et l’abondance des différentes espèces de bactéries d’un microbiome donné (Fig1.) ou les ITS ( Internal Transcribed Spacer rRNA) la variabilité intraspécifique d’un taxon . Les lectures obtenues après séquençage sont alignées sur les génomes bactériens référencés dans les bases de données pour identification et classification (taxonomie) des espèces détectées. Les séquences non assignées sont ignorées. On peut choisir une combinaison très large de barecode pour détecter un spectre d’espèces plus étendu (metabarcoding).