Genotypage (puces ADN, ARN , CpG )
Contrairement au re-séquençage de l’ADN, qui permet la détection en aveugle des variants (polymorphismes SNP, indels, LOH…) du génome, le génotypage vise à déterminer l’existence de modifications à des positions connues, sur une partie ou la totalité du génome.
Selon l’objectif du projet de génotypage plusieurs technologies sont disponibles.
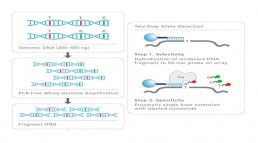
Les études d’association pangénomique, Genome Wide Association Study (GWAS), ainsi que les études de réplication nécessitent d’interroger, sur des grandes cohortes, le polymorphisme de plusieurs millions de variants repartis sur toute la longueur du génome. Les technologies à très haut débit (Illumina, Agilent, Affymetrix…), basées sur le principe de l’hybridation de l’ADN à des oligonucléotides spécifiques des variations génétiques, sont utilisées pour ce genre de projets (Fig1).
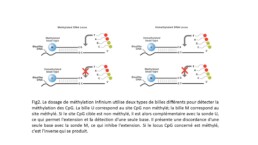
En plus des polymorphismes de séquence, il est possible, avec la même approche, de déterminer le degré de méthylation d’ilots CpG. L’ADN est préalablement traité au bisulfite qui converti les cytosines non méthylées en uracile. Le niveau de méthylation est déterminé par le ratio entre les signaux des sites méthylés vs les sites non méthylés (Fig2).
Le spectromètre Sequenom à moyen débit utilisant la technologie MALDI-TOF, Matrix-Assisted Laser Desorption/Ionisation Time of Flight, est appropriée pour les études de réplication/ validation des régions candidates issues d’un GWAS (plusieurs dizaines de variants).
Le système 7900-HT d’Applied Biosystems utilisant la chimie Taqman basée sur la discrimination allélique par amplification, est utilisé pour le « fine mapping » (génotypage de quelques variants).