Tn-Seq
Le Tn-Seq (séquençage des banques d’insertions de transposons), qui combine la mutagenèse de transposons à l’échelle du génome avec le séquençage à haut débit, permet d’assigner la fonction des gènes dans un large panel de bactéries et de procaryotes (revu dans Cain AK et al. Nat. Rev. Genet. 2020).
Le principe de base de la Tn-Seq consiste à créer une banque de souches mutantes, chacune contenant une insertion de transposon unique. Deux des transposons les plus couramment utilisés sont Himar1 de la famille mariner, qui s’insère de manière aléatoire au niveau des dinucléotides TA, et Tn5, qui peut s’insérer plus aléatoirement dans le génome. Lorsqu’un transposon s’insère dans un gène, la fonction de ce dernier est perturbée, ce qui affecte la viabilité (fitness) de la bactérie si le gène est essentiel ou a une influence sur sa croissance dans des conditions spécifiques (par exemple, exposition normale vs. exposition à des antibiotiques). Après l’enrichissement de la jonction transposon-génome consistant en une digestion par MmeI suivie d’une ligature d’adaptateur et d’une amplification par PCR, les sites d’insertion des transposons dans les souches survivantes sont déterminés à l’aide de techniques de séquençage à haut débit. En comparant la distribution des insertions dans les souches mutantes à un génome de référence, les gènes essentiels à la survie dans les conditions testées sont identifiés.
Le séquençage par insertion de transposons indexés (RB-Tn-Seq) modifie le protocole Tn-Seq initial en utilisant des transposons portant un barcode unique de 20 nucléotides, ce qui permet d’établir en parallèle le profil de fitness des mutants pour plusieurs conditions en un seul run de séquençage (Wetmore KM et al. mBio 2015).
Pour les études du génome des eucaryotes, cette technique présente certaines limites si l’on utilise le séquençage à lecture courte en raison des nombreuses régions répétées, importantes sur le plan de l’évolution ou de l’adaptation/fitness. Il en effet difficile d’établir une correspondance unique entre les reads et les séquences répétées dont les unités répétées sont plus longues que les reads séquencés. Les méthodes de séquençage à lecture longue doivent être alors privilégiées (Yasir M et al. Sci. Rep 2022).
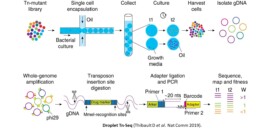
Droplet Tn-Seq (dTn-Seq) utilise des méthodes microfluidiques d’encapsulation (cf figure) pour individualiser les mutants de transposons, ce qui permet d’étudier la croissance d’une seule cellule, sans l’influence des autres populations environnantes (Thibault D et al. Nat Comm 2019).
Plateformes à contacter pour ce domaine d’expertise
Mise à jour Avril 2024