Séquençage à « longues lectures SMRT »
Basé sur la technologie SMRT (single molecule real time technology), le séquençage sur RSII et Sequel permet de produire de très longues lectures pour résoudre la couverture de régions de faible complexité telles que les régions répétées , déterminer les variants structuraux et haplotypes.
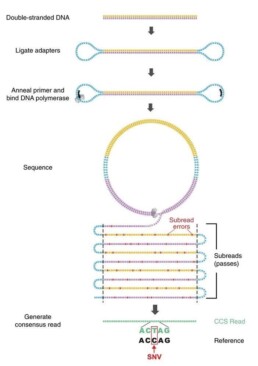
Une banque type SMRT est construite à partir de fragments de 10 à 175kb. Une fois leurs extrémités réparées, des adaptateurs simple brin y sont fixés en formant une boucle (SMRT bell), point d’ancrage pour la DNA polymérase (Figure 1).
Un complexe insert – adaptateur – polymérase est déposé et immobilisé dans chaque puits du support de séquençage, illuminé par un système ZMW (Zero Mode Waveguides) permettant la détection de signal fluorescent en temps réel (Figure 2).
La méthode de séquençage a évoluée avec la version HiFi (Wenger AM et al. Nat. Biotech. 2019). Le séquençage de l’insert circularisé n’est plus obtenu par un passage unique de la polymérase mais par des « tours » successifs. Le mode CLR (Continuous Long Read), permet d’étudier des inserts de 25 à 175kb: de multiples fragments de la même région devront être séquencés pour déterminer une séquence consensus avec une qualité de 90%. En mode HiFi, la réplication de la lecture de l’insert permet d’obtenir une séquence consensus (CCS: Circular Conscensus Sequence) de qualité (99%) à partir d’un seul fragment de 10-20kb.