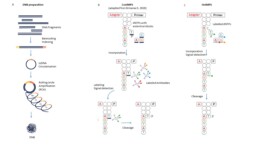
Séquençage à « courtes lectures » par DNB
La technologie proposée par MGI est basée sur le séquençage par synthèse (SBS) de pelote de DNA simple brin (DNB). La préparation des banques passe par une circularisation puis une réplication (rolling circle replication) de fragments « simple brin » de DNA/cDNA préalablement indexés pour former des DNBs de 300 à 500 copies pour chaque fragment (Figure A). Cette méthode évite les biais d’amplification et donne de faible taux de duplicats et d’erreur de séquençage.
Le séquençage par synthèse (SBS) des DNBs a évolué du système CoolMPS au HotMPS (Figure B,C)
Dans le premier cas, un dNTP portant un groupement bloquant spécifique en 3’ est incorporé à chaque cycle. Des anticorps fluorescents reconnaissant les différents groupements bloquants permettent l’identification des nucléotides. Après le clivage du complexe groupement bloquant-anticorps, un nouveau cycle peu reprendre (voir).
Le HotMPS reprend le protocole « standard » avec des « dNTP-H » qui portent des chromophores bloquants en 3’. Après identification de la base incorporée, le clivage du chromophore par une enzyme-H libère le 3’ pour une nouvelle incorporation.
La lecture du brin complémentaire (paired end) utilise le système MDA (Multiple Displacement Amplification-Dean FB 2002).