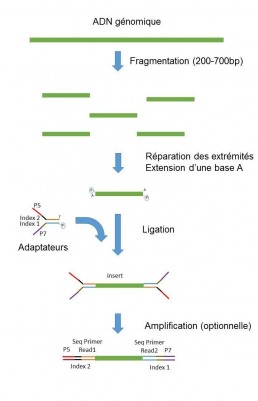
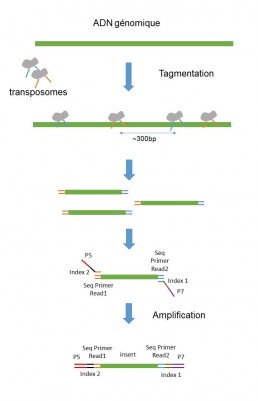
Séquençage à « courtes lectures » par clusterisation
La préparation de banques pour le séquençage en paire (« paired end »), selon la technologie lllumina, consiste à fragmenter l’ADN génomique mécaniquement (Covaris, Bioruptor) ou enzymatiquement (tagmentase) à des tailles inferieures à 1 kb.
Des adaptateurs indexés et complémentaires des amorces de séquençage, sont ensuite ligués à chaque extrémité des fragments d’ADN pour permettre le séquençage à partir de ces deux extrémités (Figure 1).
La technologie utilisant la tagmentase, une transposase modifiée, fragmente l’ADN et ajoute simultanément les adaptateurs de séquençage (Figure 2 : Technologie Nextera).
Une étape de sélection de taille sur billes peut permettre de produire une banque de fragments d’ADN d’une taille d’insert moyenne précise, ce qui peut être nécessaire pour l’assemblage de certaines régions génomiques.
Le séquençage par synthèse (SBS) commence par une étape d’amplification clonale (clusterisation) et s’effectue par une succession de cycle d’incorporation de dNTPs fluorescents, détection du signal, clivage du chromophore (voir).