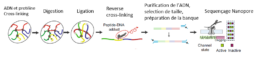
Pore-C
PORE-C est une modification du protocole Hi-C, dans laquelle le séquençage à lecture courte est remplacé par les méthodes de séquençage à lecture longue Nanopore.
Les interactions 3D d’ordre élevé entre plus de deux locus génomiques sont courantes dans la chromatine humaine, mais leur rôle dans la régulation des gènes n’est pas clair. Les précédents tests de chromatine 3D d’ordre élevé mesurent soit les interactions distantes à travers le génome, soit les interactions proximales au niveau de cibles sélectionnées. Pour combler cette lacune, Pore-C a été développé, qui combine la capture de conformation de la chromatine (3C) avec le séquençage nanopore à lecture longue de concatémères pour profiler les contacts proximaux de chromatine d’ordre élevé à l’échelle du génome [1].
Quelques méthodes générant des contacts chromatiniens multidirectionnels ont été développées, notamment la cartographie de l’architecture du génome (GAM), ChIA-drop, la reconnaissance par pool partagé des interactions par extension de balise (SPRITE), Tri-C, multicontact 4C, le test de ligature du concatémère (COLA), et Pore-C [2].
Pore-C se distingue car cette méthode peut capturer des contacts multidirectionnels globaux d’ordre élevé, est techniquement simple et capture simultanément la méthylation de l’ADN dans une population cellulaire. Étant donné que les contacts multidirectionnels reflètent des interactions synergiques de la chromatine plutôt que des interactions multiples et mutuellement exclusives de différents allèles, Pore-C peut être utilisé pour révéler une topologie à un seul allèle au sein de régions génomiques désignées dans des populations de cellules [2].
Plateformes à contacter pour ce domaine d’expertise