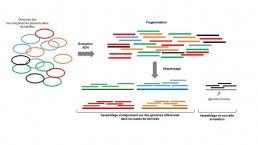
Métagénomique shotgun
Cette approche permet d’avoir un aperçu global de la composition du microbiome (bactéries, virus, champignons etc. …) ainsi que son potentiel fonctionnel par l’identification des gènes représentés. Les séquences consensus obtenues après assemblage des lectures issues du séquençage sont alignées sur des génomes référencés pour l’identification des espèces formant le microbiome. Les séquences consensus absentes des bases de données définissent des espèces nouvelles qui peuvent être à leur tour annotées et incorporées dans les bases de données (Fig1.)