Un réseau de plateformes spécialisées et complémentaires
L’infrastructure France Génomique rassemble la majorité des plateformes de séquençage et/ou de bio-informatique en France :
- les plateformes nationales du Génoscope et du CNRGH à Évry, dont les capacités de séquençage, génotypage et bio-informatique permettent la réalisation de projets à très grande échelle,
- les plateformes de séquençage ayant chacune leurs expertises et technologies spécifiques ainsi que les outils de bio-informatique ad hoc,
- les plateformes associées à France Génomique,
- le volume de données à stocker et à traiter augmentant de façon exponentiel, les partenaires de France Génomique peuvent s’appuyer sur le TGCC- Très Grand centre de Calcul- du CEA.
- FRANCE GENOMIQUE a également reconnu comme plateformes associées de bioinformatique, un certain nombre de plateformes membres de l'IFB (Institut Français de Bioinformatique), travaillant en collaboration avec les plateformes de séquençage de FRANCE GENOMIQUE et les utilisateurs de ces plateformes.
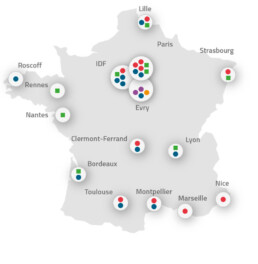
Survolez le nom de la plateforme sur la carte et cliquez pour accéder à la fiche plateforme.
- Plateformes nationales de séquençage
- Plateformes de séquençage
- Plateformes de séquençage associées
- Plateformes de bio-informatique associées
- TGCC : Très Grand Centre de Calcul
- Plateformes nationales de séquençage
- Plateformes régionales de séquençage
- Plateformes de bio-informatique
- Plateformes associées
- TGCC : Très Grand Centre de Calcul
Un parc d'équipements très haut débit et de 3ème génération
FRANCE GENOMIQUE est équipée des technologies les plus performantes et innovantes dédiées au séquençage. Le parc évolue très rapidement avec le très haut débit (NovaSeq 6000) et les appareils de séquençage dit de 3ème génération (technologie molécule unique).
FRANCE GENOMIQUE est ainsi capable de répondre à l’ensemble des demandes de projets de séquençage.
Une infrastructure de calcul haute performance
Le Très Grand Centre de Calcul (TGCC) du CEA est une infrastructure dédiée au calcul haute performance, capable d’héberger des supercalculateurs d’échelle petaflopique et conçue sur la base d’une architecture orientée vers les données. Au sein du TGCC, le CCRT dispose d’une extension qui est dédiée aux utilisateurs du projet FRANCE GÉNOMIQUE.
Cette e-infrastructure de stockage et de traitement des données, mise en œuvre par les équipes CEA/DIF permet aux utilisateurs de FRANCE GÉNOMIQUE de bénéficier d’un espace de stockage moyen terme (échelle : projets scientifiques de plusieurs années) de plusieurs petaoctets, connecté à plusieurs milliers de cœurs de calcul scalaires par une interconnexion à haute performance. Les volumes de données à stocker et à traiter augmentant de façon exponentielle, elle est également conçue pour être évolutive, avec l’objectif de relever demain l’ensemble des défis de la génomique.
Equipements et capacités
La configuration dédiée à FRANCE GENOMIQUE est composée de :
- 180 nœuds bi processeurs (Intel Sandy Bridge E5-2680, 2.7 GHz, 8 cœurs) avec 128 Go de mémoire par noeud, soit 2.880 cœurs (Bull),
- 2 systèmes à très grande mémoire Bullx S6410 à 2 To de mémoire,
- 9 lames hybrides, équipées de GPU nvidia Kepler.
L’hébergement des données est réalisé grâce à la configuration de stockage suivante :
- un stockage moyen terme présentant un système de fichiers global de 5 Po, dont 2 Po sur disque (système de stockage hiérarchique Lustre + IBM HPSS),
- un dispositif d’archivage des données initiales.
Expertise et support
Les équipes du CEA/DIF ont développé une expertise et une compétence reconnue de façon internationale aussi bien dans le domaine de la gestion des très grands volumes de données (contribution à des développements Open-source, pilotage de EOFS …) que dans la définition et le management de très grands centres de calculs. Des équipes d’assistance et de support aux utilisateurs sont disponibles pour les aider à tirer le meilleur parti des moyens du centre.
Une équipe de support applicatif dédiée est mise en œuvre par les plateformes nationales (CEA), pour le compte de FRANCE GÉNOMIQUE.
Quelques réalisations
Afin de caractériser un ensemble de 83 familles protéiques sans fonctions connues et regroupant quelques 60.000 séquences, les chercheurs de Genoscope ont mené une campagne de modélisation sur le supercalculateur Titane du CCRT. Cette phase, qui aurait nécessité 280.000 heures de calcul, a pu être exécutée en seulement 70 heures sur 4.000 processeurs. A partir des résultats, les chercheurs ont créé un catalogue de signatures structurales spécifiques pour chacune des familles étudiées. Ce catalogue va apporter aux biochimistes des informations précieuses pour découvrir de nouvelles activités enzymatiques.
Le Genoscope utilise les moyens de calculs du TGCC/CCRT depuis déjà plusieurs années, notamment via les appels à projets DARI. Dans ce cadre, le projet TARA OCÉANS a bénéficié de plus de 3,5 millions d’heures de calculs pour étudier la diversité des organismes marins. Pour ce faire, différents outils d’analyse de séquences ont été portés sur cette infrastructure : BLAST, BLAT, InterProScan & CDDsearch. Des codes spécifiques ont été conçus et déployés afin d’adapter ces outils aux contraintes techniques d’exploitation des machines du TGCC (parallélisation massive par les données, contrôle d’exécution, reprise sur erreur, jobs unitaires courts).
En savoir plus
Site web : www-hpc.cea.fr/, www-ccrt.cea.fr
Responsable de la plateforme : Pierre Leca
CEA DAM-île de France
Bruyères-le-Châtel
91297 Arpajon Cedex
Contact: e-infrastructure@france-genomique.org

Illumina propose du séquençage de haut débit et très haut débit.
Après amplification clonale de fragments d’ADN courts, le séquençage par synthèse (SBS) commence : chaque base émet un signal de fluorescence unique lorsqu’elle est ajoutée au brin en cours de synthèse. La détection du signal à chaque incorporation détermine la séquence d’ADN.
La large gamme d’appareils permet de répondre à tout un panel de besoins tant en terme d’applications ou de rendement.

Les instruments de séquençage d’ADN de MGI utilisent la technologie de base appelée DNBSEQTM.
Les DNB (nanobilles d’ADN) sont pompées par le système fluidique et chargés sur une puce gravée de motifs.
L’amorce de séquençage est ensuite ajoutée et hybridée à la région adaptatrice du DNB. La réaction de séquençage commence par le pompage de réactifs de séquençage contenant des sondes dNTP marquées par fluorescence et de l’ADN polymérase. Les images sont prises après que les sondes marquées par fluorescence sur le DNB soient excitées avec des lasers. Les images sont ensuite converties en un signal numérique. Cette information est ensuite utilisée pour déterminer la séquence d’ADN de l’échantillon.

La plateforme de séquençage PacBio est une plateforme de séquençage à lecture longue.
La technologie de base, le temps réel à molécule unique (SMRT), permet de générer des lectures d’une longueur de plusieurs dizaines de kilo-bases. Le séquençage SMRT évite largement les biais spécifiques à la séquence dans le système NGS, dans la mesure où la plupart des étapes d’amplification PCR ne sont pas requises dans le processus de construction de la banque.
Oxford Nanopore Technologies propose une technologie de séquençage de l’ADN et de l’ARN en temps réel sans synthèse et sans amplification, la lecture s’effectuant au travers d’un nanopore soumis à un champ électrique.
Le courant ionique diffère selon la base A, T, G ou C qui obstrue le nanopore. L’identification de la séquence se fait par la mesure de l’évolution du courant ionique traversant le nanopore.

Le séquenceur AVITI d’Element Biosciences permet un séquençage à moyen débit avec une qualité supérieure et des coûts inférieurs comparés aux séquenceurs Illumina.
Bien que la chimie de séquençage soit très différente, l’instrument est compatible avec les banques Illumina. Contrairement à la chimie de séquençage par synthèse (SBS) traditionnelle, l’AVITI utilise une chimie de séquençage par liaison (SBB) qui nécessite la liaison d’un substrat de polymérase fluorescente multivalente par « avidité ». L’utilisation de ces avidites augmente la spécificité et réduit les coûts de la chimie fluorescente d’un ordre de grandeur. Grâce à cette technologie, les étapes de synthèse de l’ADN peuvent être réalisées avec des nucléotides non marqués.

La société 10x Genomics a développé une machine permettant de résoudre en partie les écueils du séquençage par synthèse (SBS) à lectures courtes: le Chromium.
Ce système utilise une méthode de PCR en émulsion. La création de l’émulsion a pour rôle d’encapsuler dans une goutte de liquide réactionnel quelques molécules d’ADN de haut poids moléculaire dans le cas de séquençage de longue lecture synthétique, ou une cellule dans le cas de séquençage « cellule unique ».
Cette méthode permet de faire des assemblages de lectures courtes (Illumina) via un système de barcoding unique, rendant accessibles des informations à longue distance, ce qui facilite l’analyse du phasage et la caractérisation de structures chromosomiques. Il permet également d’étudier le transcriptome par RNAseq de plusieurs milliers de cellules uniques en parallèle.
Après préparation des librairies, le séquençage s’effectue sur machine Illumina.