Enzymatic Methyl-seq (EM-seq™)
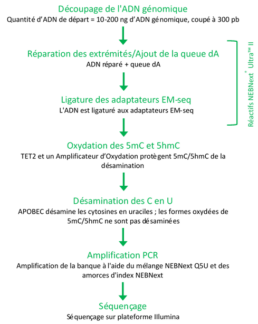
Le séquençage au bisulfite détecte la 5-méthylcytosine (5mC) et la 5-hydroxyméthylcytosine (5hmC) à la base près. Cependant, le traitement au bisulfite endommage l’ADN, ce qui entraîne une fragmentation, une perte d’ADN et des données de séquençage biaisées. Pour pallier à ces problèmes, la méthode méthyl-seq enzymatique (EM-seq™) a été développée par NEB, voir image 1 (NEBNext® Enzymatic Methyl-seq Kit, site NEB).
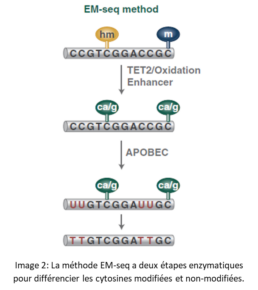
Cette méthode détecte 5mC et 5hmC en utilisant deux réactions enzymatiques (image 2).
Dans la première réaction, TET2 (Ten-eleven-translocation 2 = méthylcytosine dioxygénase) et T4-BGT (T4 Phage β-glucosyltransférase) convertissent 5mC et 5hmC en produits qui ne peuvent pas être désaminés par APOBEC3A (enzyme d’édition d’ARNm de l’apolipoprotéine B, qui se comporte comme un polypeptide catalytique ; APOBEC désigne une famille de cytidine désaminases, conservées de manière évolutive). Dans la deuxième réaction, APOBEC3A désamine les cytosines non modifiées en les convertissant en uraciles. Ces trois enzymes permettent donc l’identification de 5mC et 5hmC (Vaisvila et al., 2021).
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- TAPS/TAPSβ
- Méthylation de l’ADN et ARN Natifs
- Cartographie des sites ADN – Protéines de la chromatine : CUT & RUN vs CUT & Tag
- Cartographie des contacts de la chromatine : 3C, 4C et 5C
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie des sites d’accessibilité de la chromatine : DNase-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : FAIRE seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq
- BiSeq