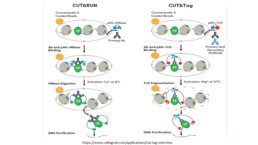
Cartographie des sites ADN – Protéines de la chromatine : CUT & RUN vs CUT & Tag
CUT & RUN (Cleavage Under Target & Release Using Nuclease) fait partie de la série de protocoles (CHIP, ATAC, FAIRE seq….) utilisés pour cartographier les nucléosomes et l’accessibilité de la chromatine pour les facteurs de régulation de la transcription . Cette nouvelle méthode a l’avantage de s’opérer in situ, sur cellules ou noyaux intacts, en s’affranchissant des étapes de crosslinking et de fragmentation qui produisent des fragments d’ADN en dehors/éloignés des sites d’interactions ADN protéines créant beaucoup bruit de fond de séquençage. Selon la spécificité de l’anticorps, CUT & RUN permet de cibler les modifications des histones, les facteurs de transcriptions et cofacteurs (PJ Sken and S Henikoff, 2017).
La méthode CUT & RUN consiste schématiquement à immobiliser des cellules perméabilisées ou des noyaux sur des billes magnétiques et à les incubés en présence d’anticorps spécifiques des protéines cibles (modifications d’histones , facteurs de transcription, etc. ) puis de Protéine A-MNase qui diffusent par les pores du noyau. Une fois l’ancrage cible-anticorps – Proteine A-MNase établi, du Ca++ est ajouté à basse température pour activer Protéine A-MNase et cliver l’ADN aux 2 extrémités du complexe ADN-protéines cibles . Les particules sont récupérées et l’ADN est purifié pour entrer dans le protocole classique de préparation de banques pour le séquençage. Environ 3 à 5M de lectures sont suffisants pour l’analyse ( près de 1/10 de la couverture nécessaire pour le CHIP seq)
L’efficacité de la méthode dépend de la spécificité de l’anticorps et de l’abondance de la cible .
Ce protocole a été adapté pour les études sur cellules uniques et noyaux avec le CUT & Tag (Kaya-Okur, H.S. et al. 2020) qui utilise la pA-Tn5 qu’on peut indexer avec des adaptateurs. Son activation en présence de Mg++ entraine la tagmentation avec intégration des adaptateur dans l’ADN cible et ainsi évite l’étape de ligation dans la préparation de la banque pour le séquençage (voir figure)
Plateformes à contacter pour ce domaine d’expertise
Mise à jour : Avril 2024
Rubriques associées
- Séquençage des petits ARN
- Cartographie des sites d’initiation de la transcription : TSS seq
- TAPS/TAPSβ
- Enzymatic Methyl-seq (EM-seq™)
- Méthylation de l’ADN et ARN Natifs
- Cartographie des contacts cis et trans de la chromatine : HiC-seq
- Cartographie indirecte des sites d’accessibilité de la chromatine : MNase seq
- Cartographie des sites d’accessibilité de la chromatine : ATAC seq
- Cartographie des sites des interactions ARN- protéines : CLIP seq
- Cartographie des sites des interactions ADN- protéines : CHIP seq
- Cartographie des marques épigénétiques de l’ADN : MeDIP
- Cartographie des marques épigénétiques de l’ADN : Methyl seq
- BiSeq